Un exemple de simulation
Chercheur : Benjamin Bouvier, laboratoire LG2A de l'UPJV et CNRS Domaine : dynamique moléculaire
La dynamique moléculaire est un outil essentiel pour comprendre à l’échelle atomique les mécanismes de l’interaction entre les biomolécules responsables de la vie cellulaire, notamment à des fins thérapeutiques.
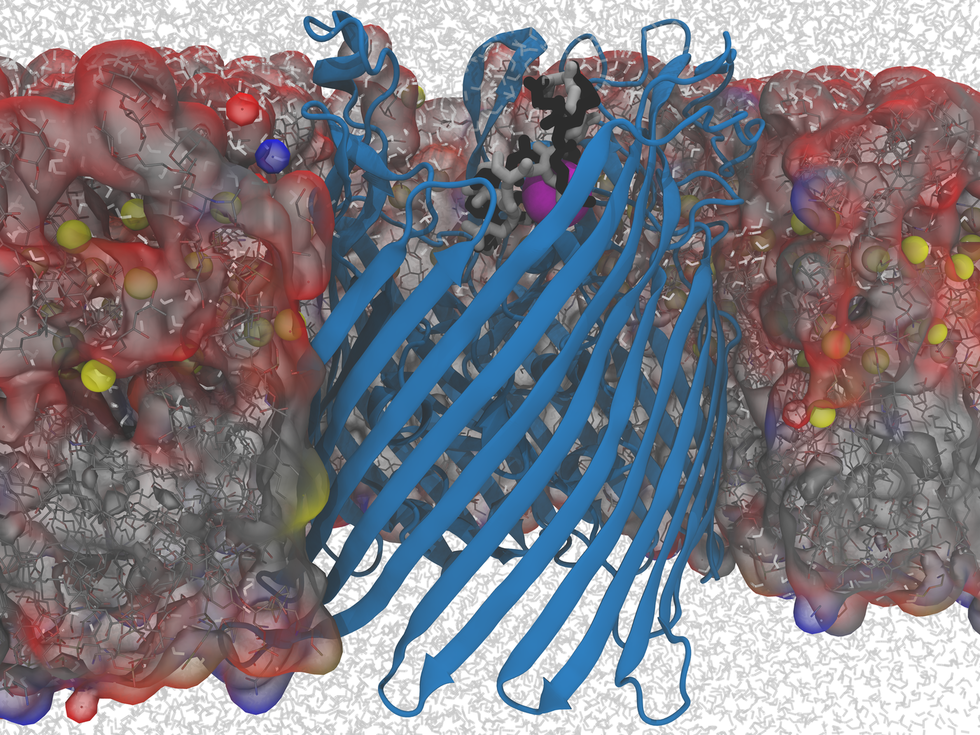
Pseudomonas Aeruginosa est une bactérie responsable d’infections nosocomiales. Elle récupère dans le milieu extérieur le fer nécessaire à sa survie en y secrétant la pyoverdine, une molécule capable de piéger cet élément puis d’être réincorporée dans la bactérie par des transporteurs membranaires spécifiques. Nous avons étudié par dynamique moléculaire les mécanismes de la reconnaissance entre la pyoverdine (bâtonnets gris sur l’image) liée au fer (sphère violette) et ces transporteurs membranaires (bleu), afin de concevoir des analogues de pyoverdine (bâtonnets noirs) capables à la fois d’être reconnus par ces derniers et d’en bloquer irrémédiablement le fonctionnement, conduisant à la mort de la bactérie.
Les calculs sur ce système, qui comprend 150 000 atomes, ont nécessité environ 400 000 heures-CPU dont plus d’un quart a été mené sur la plateforme MeCS.
